导语
目前16s RNA已经被广泛应用到对微生物多样性的评估当中,但是我们一般都会遇到一些问题,我们该怎么解决?一起来看看。
问题:
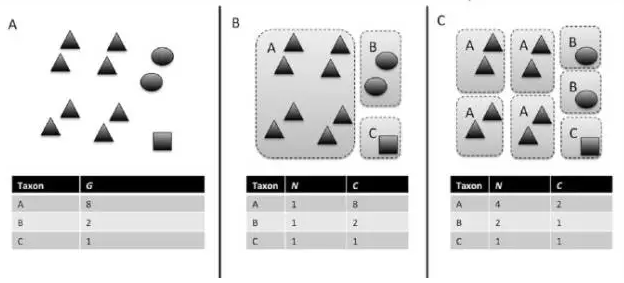
G 代表测序中某个分类对应的reads
N 代表某个分类表达量
C 代表copy数目
所以说copy数目,对最后微生物多样性的评估影响比较大,而根据目前已知的16s RNA copy数目从1-15之间从在较大的差异性,因此为了弥补这样的差异性,一般可以寻找单copy的新的分子标记物,例如ropB基因等等。但是这样的分子又没有被广泛使用,所以对于后期的处理也带来了一定的难度。
为了解决以上的问题,我们仍然采用16s RNA作为生物分子学标记,然后融入copy信息,主要是借助进化树的相关方法:
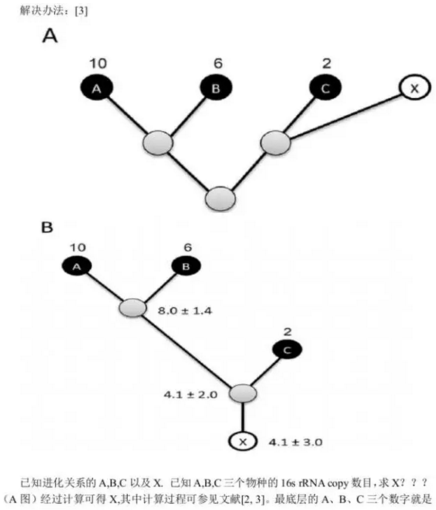
通过copy数字主要来自rrnDB database [1]
具体到实现过程:
1:测序数据(sequence reads)
2: Aligned and masked to the same Greengenes core data set as the reference taxa using PyNAST
3:建立进化树(pplacer)
4:使用文献[3]提供的R脚本,建立合理的16s rRNA物种丰度表达
参考文献:
1. Klappenbach, J.A., et al., rrndb: the Ribosomal RNA Operon Copy Number Database.Nucleic Acids Research, 2001. 29(1): p. 181-184.
2. Case, R.J., et al., Use of 16S rRNA and rpoB Genes as Molecular Markers for Microbial Ecology Studies. Applied and Environmental Microbiology, 2007. 73(1): p. 278-288.
3. Kembel, S.W., et al., Incorporating 16S Gene Copy Number Information Improves Estimates of Microbial Diversity and Abundance. PLoS Comput Biol, 2012. 8(10): p. e1002743.
特别声明:本文来源于fanyucai的博客,仅仅是出于传播信息的需要,作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。