Transcriptomic basis for drought resistance in Brassica napus L
基于转录组研究甘蓝型油菜的抗旱性
Pei Wang, Cuiling Yang, Hao Chen, Chunpeng Song, Xiao Zhang & Daojie Wang
期刊:Scientific reports ,2017.01,IF=5.1
研究背景:油菜为异源四倍体植物,是芸薹(B. rapa )和 甘蓝(B. oleracea)通过自然种间杂交后双二倍化而来的一种复合种。为世界第三大产油蔬菜。干旱是非生物胁迫因子严重影响植物生长和农业生产。培育抗旱和抗病品种是育种的长期目标。鉴定抗旱基因和评估这些基因的潜在功能适应胁迫非常重要。目前,关于油菜抗旱相关基因的研究非常少,本文旨在探索鉴定和分析油菜的抗旱相关基因。
材料:不同类型的两个油菜抗旱品种07Y19(抗旱:RT)和07Y29(不抗旱:ST),实验组:将这两个品种的种子用200 g L−1 PEG-6000处理后(标记为 R和 S),对照组: RT和ST相同环境下栽培不做任何处理。实验组测叶片的失水率衡量干旱胁迫。PEG-6000处理后分8个时间点取样提取RNA: 0.5 h, 1 h, 3 h, 6 h, 9 h, 12 h, 24 h ,48 h。 每个样品包含10个个体。
测序策略:Illumina HiSeq™ 2000,PE100,5G data
研究思路:
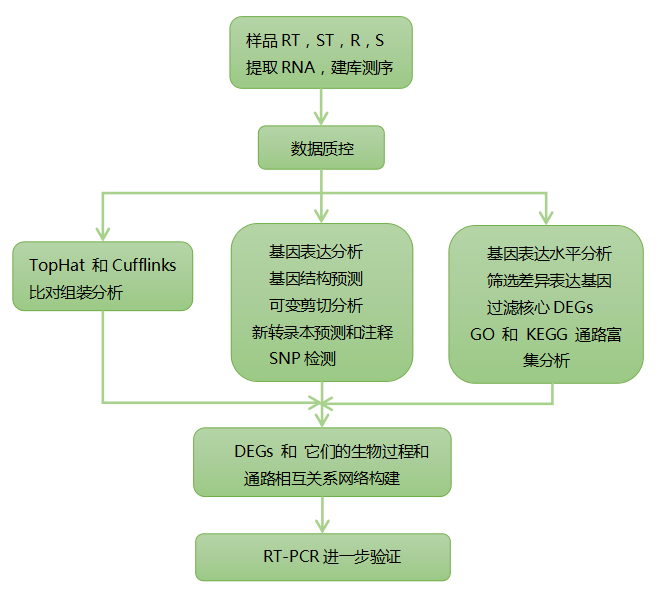
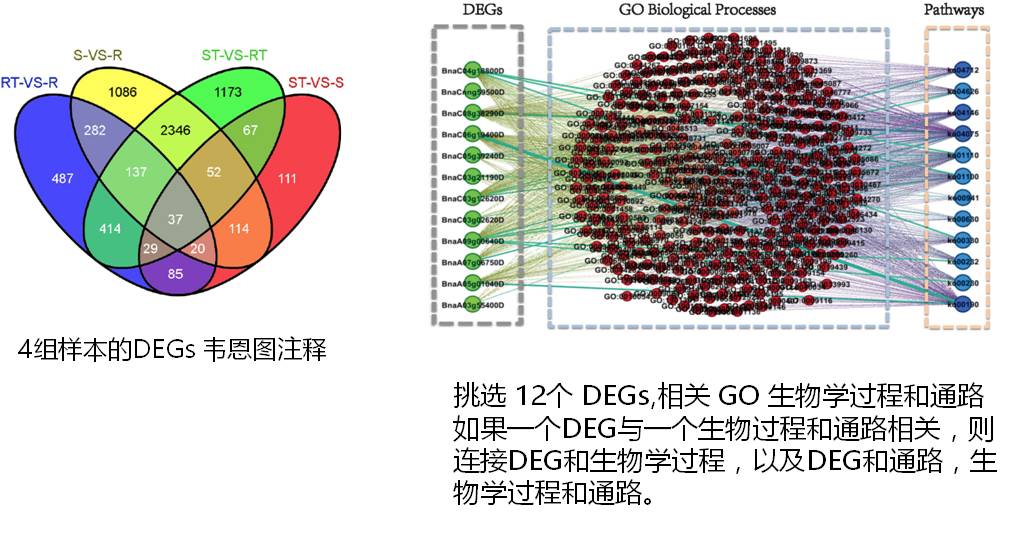
研究结果:本文将抗旱和不抗旱的油菜(Brassica napus L)在干旱和水分充足的条件下栽培后进行转录组测序,统计学分析有3类包含169个显著差异表达基因(DEGs)与干旱相关。包括37个抗旱品种相关基因,35个不抗旱的基因,97个特性不明显的基因。这些DEGs相当均匀分布在不同的染色体上,它们的表达模式有品种特异性。
除了一般在响应不同的刺激或压力时富集,不同类别的DEGs在特定的生物过程或通路中特异富集,比如:多种刺激,代谢过程,代谢通路,次生代谢物的生物合成,淀粉和蔗糖代谢,苯丙烷生物合成,玉米素合成。抗旱品种中的DEGs还参与植物信号转导,ABC转运蛋白和过氧化磷酸化途径。而类黄酮生物合成途径特异存在于不抗病品种中。GO和KEGG通路分析证明这些核心DEGs参与一些功能范畴与干旱胁迫相关,以及相关的通路和过程在响应干旱胁迫时发挥着重要作用。表明这三类核心DEGs之间的功能可能有差异。
构建网络图显示这169个DEGs之间的关系,生物学过程和通路。根据最可靠的途径和生物学过程将这些DEGs分为功能不同的类别。一些通路可能同时有多个共同的DEGs参与,这些通路可能相通以及有重叠的生物学功能。少数DEGs能适应拟南芥的干旱胁迫信号转导途径。最后用QT-PCR验证了RNA-seq数据的准确性。这些研究对通过转基因工程改善作物品种有一定的作用。
模式植物水稻,玉米,拟南芥中抗旱相关的研究
 In 2009, Degenkolbe et al.研究了两个水稻抗旱品种和不抗旱品种在干旱胁迫下的基因表达谱 在这四个品种中,仅有245个基因下调,413个基因上调。
In 2009, Degenkolbe et al.研究了两个水稻抗旱品种和不抗旱品种在干旱胁迫下的基因表达谱 在这四个品种中,仅有245个基因下调,413个基因上调。
 In 2011, Liang et al.利用消元法预测出了拟南芥抗旱相关基因,基于CEO数据库(http://www.ncbi.nlm.nih.gov/geo/)中22套拟南芥的基因表达数据,并且检测出靠前7个基因与抗旱生物进程相关。
In 2011, Liang et al.利用消元法预测出了拟南芥抗旱相关基因,基于CEO数据库(http://www.ncbi.nlm.nih.gov/geo/)中22套拟南芥的基因表达数据,并且检测出靠前7个基因与抗旱生物进程相关。
 In 2012,Kakumanu et al. 应用转录组测序研究了玉米再生组织和叶片分生组织中干旱对基因表达的影响。发现抗旱相关基因在玉米子房中多于叶片分生组织。
In 2012,Kakumanu et al. 应用转录组测序研究了玉米再生组织和叶片分生组织中干旱对基因表达的影响。发现抗旱相关基因在玉米子房中多于叶片分生组织。
油菜抗性相关的一些研究
 2014, Chalhoub et al.1首次发表了完整的油菜基因组序列, 但是基于组学水平的研究仍然很少。
2014, Chalhoub et al.1首次发表了完整的油菜基因组序列, 但是基于组学水平的研究仍然很少。
 In 2014, Yong et al. 通过RNA-Seq,对油菜B. napus叶片和根响应高盐环境进行了比较转录组分析,在响应盐激时,总共有582个转录因子和438个运输机因调节两个组织,鉴定出几个重要通路参与盐胁迫转导。
In 2014, Yong et al. 通过RNA-Seq,对油菜B. napus叶片和根响应高盐环境进行了比较转录组分析,在响应盐激时,总共有582个转录因子和438个运输机因调节两个组织,鉴定出几个重要通路参与盐胁迫转导。
近来, Zhang et al. 通过全基因组关联研究(GWAS),鉴定出16个基因座与水分胁迫显著相关。结合由RNA-seq检测出的DEGs和GWAS分析出的基因座,作者鉴定出了79个候选基因在油菜忍耐水分胁迫时起重要作用,其中有8个基因可能与抗干旱相关。
油菜相关参考文献
1. Sahni, S., Prasad, B. D. & Liu, Q. Overexpression of the brassinosteroid biosynthetic gene DWF4 in Brassica napus simultaneously increases seed yield and stress tolerance. Sci Rep 6, 28298 (2016).
2. Wu, J., Zhao, Q. & Yang, Q. Comparative transcriptomic analysis uncovers the complex genetic network for resistance to Sclerotinia sclerotiorum in Brassica napus. Sci Rep 6, 19007 (2016).
3. Liang, Y., Xiong, Z. & Zheng, J. Genome-wide identification, structural analysis and new insights into late embryogenesis abundant(LEA) gene family formation pattern in Brassica napus. Sci Rep 6, 24265 (2016)
4. Wang, D. et al. Comparative transcriptome analyses of drought-resistant and -susceptible Brassica napus L. and development of EST-SSR markers by RNA-Seq. J Plant Biol 58, 259–269 (2015).
5. Yong, H. et al. Comparative transcriptome analysis of leaves and roots in response to sudden increase in salinity in Brassica napus by RNA-seq. BioMed Res Int 2014, 467395 (2014).
6. Zhang, J. et al. Identification of Putative Candidate Genes for Water Stress Tolerance in Canola (Brassica napus). Front. Plant Sci. 6,1058 (2015).
7. Jiang, J. et al. Use of digital gene expression to discriminate gene expression differences in early generations of resynthesized Brassica napus and its diploid progenitors. BMC Genomics 14, 72 (2013).