【中英文题目】
An evaluation of thePacBio RS platform for sequencing and de novo assembly of a chloroplast genome
通过对叶绿体基因组的测序及从头组装对PacBio RS测序平台进行评估
【基本信息】
期刊:BMC GENOMICS
IF:4.276
年份:2013
【摘要】
背景:二代测序技术已经可以为越来越多的非模式生物在全基因组水平上描述序列特征,但是,测序读长较短,基因覆盖区具有偏向性,后期组装繁琐。PacBio RS测序平台增加了reads长度,基因覆盖区无偏向性,因此,最终产生的基因组序列就拥有较少的gap和较长的contig。但是,三代测序的缺点是成本和错误率都较高。本次研究通过对Potentilla micrantha(委陵菜甘菊)叶绿体基因组的测序以及从头组装从而对PacBio RS测序平台进行评价。
结果:从叶绿体基因组中一共得到28,638个PacBio RS reads,每个reads的平均长度为1,902bp, 测序深度为320×。对于单个contig,PacBio RS测序数据完全覆盖了叶绿体基因组的154,959bp (100% coverage),相比Illumina七个contig(90.59% coverage),而且,对于GC富集区域也并没有明显偏好性。后期序列的组装与Illumina类似,允许在两端的反向重复区域存在一些差异。
结论:本次研究是基于叶绿体基因组PacBio测序数据进行从头组装的第一次报道,用来组装的PacBio数据只产生一个较大的contig,与Illumina相比,产生的reads较长并且具有较低的GC偏好性。研究表明,PacBio测序对于基因组研究具有很大的实用性,相比Illumina产生的短reads,它并不会产生很多gap和contig.
【研究思路】
取材:
Potentilla micrantha(以下简称P. micrantha)取自塞尔维亚的阿瓦拉山,将其带回实验室并在适宜条件下培养,促进其快速生长,之后,提取叶片DNA.
文库构建:
分别利用PacBio RS和Illumina HiSeq2000对提取到的DNA进行建库
测序策略:
Pacific Biosciences PacBio RS利用单分子实时(SMRT)测序技术 ;HiSeq 测序系统既有Illumina和Solexa在边合成边测序上的优势,又融合了最新的光学图谱。
信息分析:
使用AbySS默认参数对Illumina测序数据进行拼接及组装 |
PacBio RS reads错误纠正、叶绿体reads提取以及序列组装 |
利用SMRT的HGAP(分层基因组组装过程)对测序数据进行纠正 |
利用Celera Assembler将PacBio测序数据进行组装 |
绘出基因组覆盖图,同时,分别绘制出这两组数据的GC含量图并计算相关系数 |
计算PacBio和Illumina数据集的相对误差率 |
基因注释以及与Fragaria vesca(野草莓)叶绿体基因组的比较 |
Pacific Biosciences PacBio RS |
【研究结果】
1.Illumina HiSeq2000和PacBio RS的测序数据
在进行误差纠正之前,提取只包含叶绿体基因组的序列,PacBio RS reads的平均长度是3,936.66 bp, 一共含有223,483,907个核苷酸,经过HGAP误差纠正之后,还有28,638个PacBio RS reads平均长度为1,902.75 bp且一共含有54,492,250 bp核苷酸。经过修剪以后,Illumina reads一共含有7,164,496对,平均长度在99.22bp,一共含有核苷酸1,421,726,349个。
2.叶绿体基因组的组装
PacBio RS与Illumina HiSeq2000所产生的数据以及序列的组装的比较见图1与表1
表1 P. micrantha叶绿体基因组的序列的统计
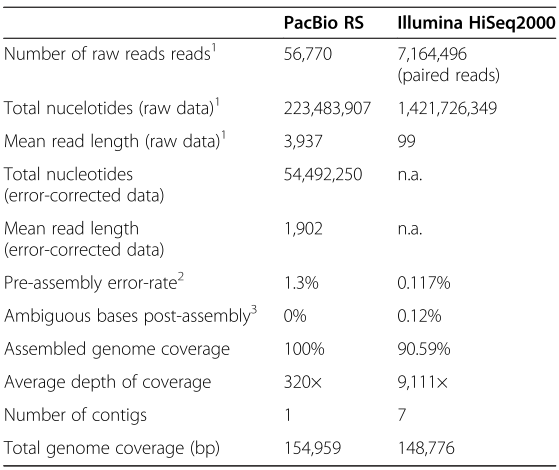
使用PacBio RS和Illumina HiSeq2000得到的P. Micrantha叶绿体基因组的测序数据汇总统计
1表示修建的Illumina reads
2表示误差修正的PacBio reads和原始的Illumina reads
3表示与叶绿体一致性序列的比较
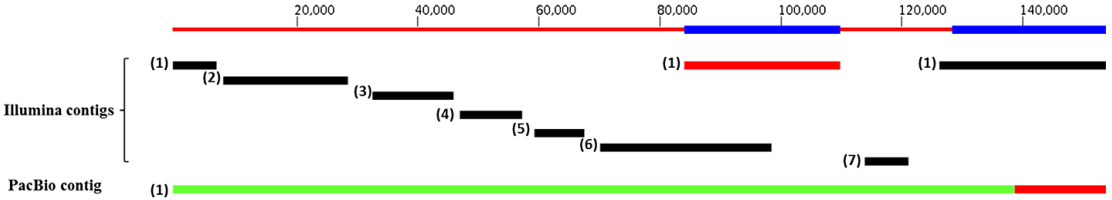
图1 P. micrantha叶绿体基因组的序列覆盖范围 示意图显示的是将P. micrantha叶绿体基因组通过ABySS和Celera assembler组装得到的Illumina(黑色)和PacBio(绿色)contigs. 示意图最上面的红色的线条表示的是叶绿体基因,蓝色加粗的区域表示的是基因组中反向重复的区域,Illumina和PacBio组装得到的contig 1中并非IR唯一的部分用红色表示出来
3.覆盖深度和GC偏好性
PacBio和Illumina reads分别覆盖了P. micrantha叶绿体基因组的100%和99.6%的,Illumina组装后含有低覆盖度的区域(图2),意味着7个contigs只是覆盖了叶绿体一致性基因组的90.59%(图1);PacBio数据则明显更为均匀(图2),并且仅仅组装成一个contig, 也是形成叶绿体一致性序列的基础(图1)。图2展示的是PacBio和Illumina在叶绿体基因上的每个碱基的覆盖度,表明PacBio RS的测序结果表现出更加均匀的覆盖度。
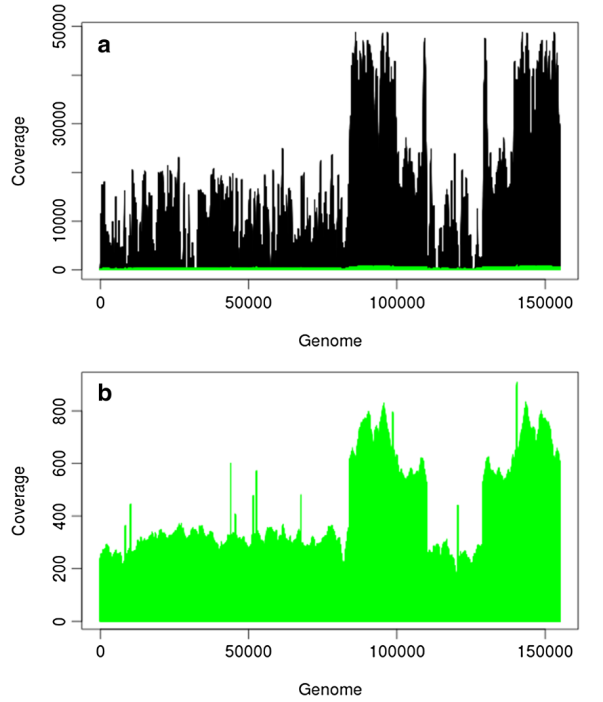
图2 P. micrantha叶绿体基因组序列每个碱基的覆盖范围 图中显示的是叶绿体基因组的每个碱基之间的测序深度,(a)表示Illumina (黑色)和PacBio (绿色)测序数据 (b)表示只有PacBio测序数据,反映出的是PacBio测序数据尽管大大降低了覆盖度,但是在基因组上的覆盖相对均匀,对于Illumina测序数据,还含有很低甚至是0覆盖度。此外,两个数据集中显著较高覆盖度的是反向重复序列。
为了检测GC偏性是否存在于这两类测序结果中,文章对平均覆盖度和GC百分含量做了相关性分析,在计算的过程中,排除了反向重复序列。计算得到的PacBio和Illumina数据集的皮尔森系数分别是0.23 (p-value = 5.675e-09)以及0.61 (p-value = 2.2e-16),因此,Illumina数据集中平均覆盖度和GC百分含量表现出很强的相关性(图3)。
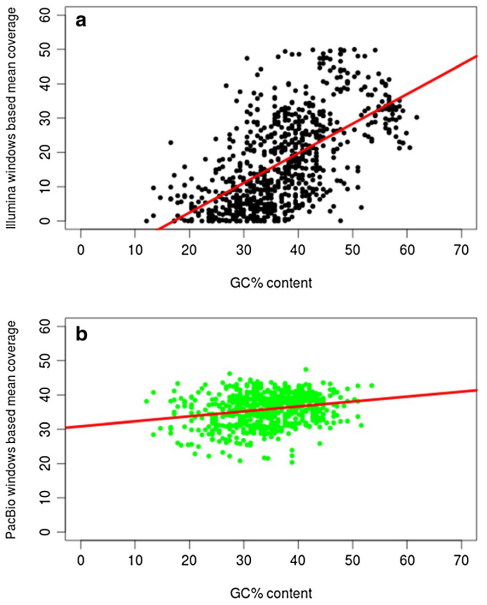
图3 Illumina and PacBio测序数据集的GC偏性的计算 分别根据(a) Illumina(黑色)(b) PacBio(绿色)测序数据中,含157个核苷酸的987个窗口的平均测序深度,计算GC含量并作图,结果表明,Illumina数据(皮尔森相关系数= 0.61,p值 = 2.2e-16)比PacBio数据(皮尔森相关系数= 0.23,p值 = 5.675e-09)显示出与GC含量的更强的相关性,排除反向重复序列的高覆盖度数据。
4.错误率
组装前的PacBio RS reads与P. micrantha叶绿体基因组一致性序列相比较,其错误率为1.3%,而Illumina reads的错误率仅是0.117%.
5.叶绿体基因组针对不同测序深度的组装
为了了解使用PacBio RS测序数据时,测序深度对于P. micrantha基因组组装的影响,使用10×, 20×, 35×, 50×,100×, 150×和200×的样本数据,将每个数据集进行组装,7个组装装好的数据集中,有5个(从 200× 到35×)得到的是单个的contig, 同时,测序深度为20× 的数据集得到4个contigs覆盖基因组的95.6%,测序深度为10×的数据集得到的是14个contigs覆盖基因组的78.2%. 为了进行比较,Illumina测序数据取与PacBio相同的7个测序深度的样本数据,并进行组装,但是,与9111×测序深度相比,并没有得到比较完整的组装。
6. P. micrantha叶绿体基因组的结构
组装好的P. micrantha叶绿体基因组长154,959 bp(图4), 反向重复序列(IR) 25,530 bp, 大的单一重复区(LSC)和小的单一重复区域(SSC)分别长85,137 bp和18,762 bp. P. micrantha叶绿体基因组共包含了120个基因,有141个基因功能已知。其中,31个是tRNA编码基因,7个位于IR区。
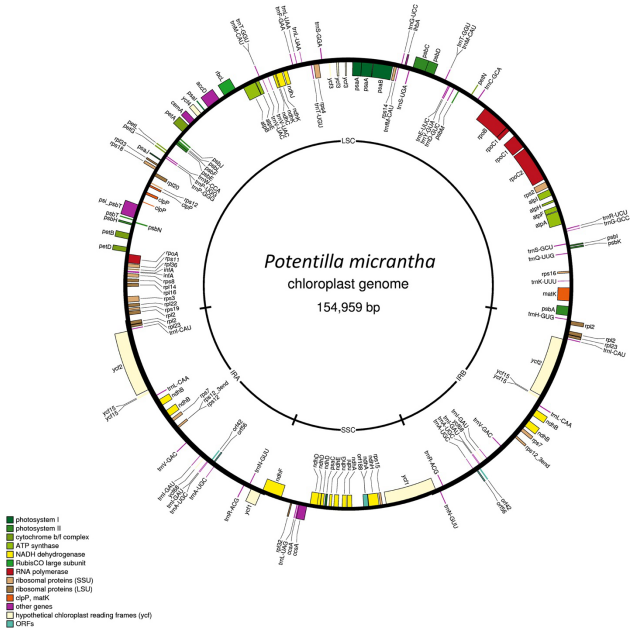
图4 P. micrantha叶绿体基因组序列 P. micrantha叶绿体基因组基因含量的结构图,外圈内侧表示基因顺时针转录,外圈外侧表示基因逆时针转录,根据不同的功能将基因带有不同的颜色;内圈表示基因组的平均GC含量。IRa和IRb表示反向重复区域,LSC和SSC分别表示长重复区域和短重复区域。基因图谱经OGDRAW绘制。
【研究结论】
1、 文章第一次报道了使用PacBio RS测序平台对叶绿体基因组的从头测序与组装,为了更好地评价PacBio RS测序结果,文章还将其与Illumina HiSeq2000测序平台的测序结果进行了比较。
2、 研究表明,PacBio测序对于基因组研究具有很大的实用性,相比Illumina产生的短reads并且具有GC偏好性低,它并不会产生很多gap和contig.
3、 PacBio RS测序技术有利于完成不同真核生物高质量的测序。
【所用软件及数据库】
SMALT:序列比对工具,文中用来过滤和提取叶绿体DNA reads
AbySS:序列的拼接与组装是基因组测序数据处理中一个至关重要的步骤,AbySS是用于高通量测序序列拼接与组装的软件
CD-Hit:通过序列比对聚类(Cluster)的方法去除冗除、相似的序列,最后输出一个非冗除(non-redundant,nr)的序列文件
SMRT:Pacific Biosciences公司的SMRT技术,作为第三代测序技术,与前两代技术相比,其最大的特点是单分子测序
DOGMA:基因组注释工具
OGDRAW:在基因组学研究中,用于画一个小的环形基因组,例如线粒体、叶绿体、质粒的圈图
CORAL:纠正下一代测序数据错误率的工具