【基本信息】
期刊:SCIENTIFIC REPORTS
IF:5.525
年份:2016
【摘要】
鮸仔(miiuy croaker),鮸鱼属,作为黄花鱼的代表,以它的特别大的耳石而著名。这种生物主要栖息在含有泥沙的浑浊的水中,但是这种生物的免疫系统和特定水生环境的特征并没有很深入的研究,因此,研究者做了鮸仔的高质量的基因草图,发现几个对鱼先天免疫至关重要的基因家族;和其他鱼的基因组相比较,鮸仔的感官系统还是发生了一些变化,包括视力的改变以及味觉和嗅觉受体的改变,这些改变使得鮸仔在长期的自然选择中可以适应环境的变化。鮸仔的基因组研究可以为它的发达的免疫系统以及在浑水中生存的适应模式提供可靠信息。
【研究思路】
取材:
野生的雌性鮸仔取自中国浙江省的东海区域并提取腹肌的DNA进行测序
文库构建:
根据Illumina标准实验计划建库(包括7个短插入序列的双端测序库和4个长插入序列的配对测序库)
测序策略:
使用Illumina Hiseq2000测序,产生了101bp或151bp的reads,经过筛选过滤后,剩下的进行从头组装。
信息分析:
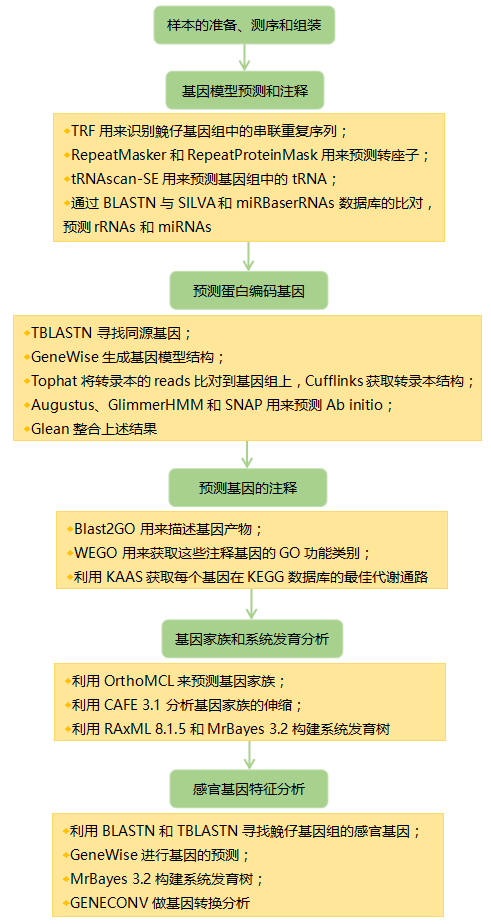
【研究结果】
1. 对鮸仔的系统发育及进化分析
为了确定鮸仔在系统进化过程中的位置,文章将其与其他20种脊索动物(包括11种硬骨鱼teleosts)进行比较,使用所有物种共有的560个同源基因构建系统发育树。根据图1A的系统发育树显示,刺鱼(stickleback)是和鮸仔关系最近的物种,刺鱼和鮸仔的旁系群是魨形目(Tetraodontiformes);对鮸仔以及三个典型的硬骨鱼种进行聚类分析,发现在这四个物种中,共有9,121个保守的基因家族(图1B)。通过对基因簇的比较发现,在所有研究的物种中,有5,780个保守的基因家族(图1C),同时,在鮸仔中有1,208个基因家族发生扩增这些扩增的基因家族主要参与钙离子的结合作用,眼睛的形态发生作用以及肌细胞的发育作用(图1C)。这些基因家族的扩增可能与一些基本的生命活动相关,比如钙的代谢作用、视觉和肌肉细胞的生长发育过程。另外,30个主要缩小了的基因家族包括MHC(在获得性免疫中起到主要作用),这也说明了鮸仔的获得性免疫的不完全发育。
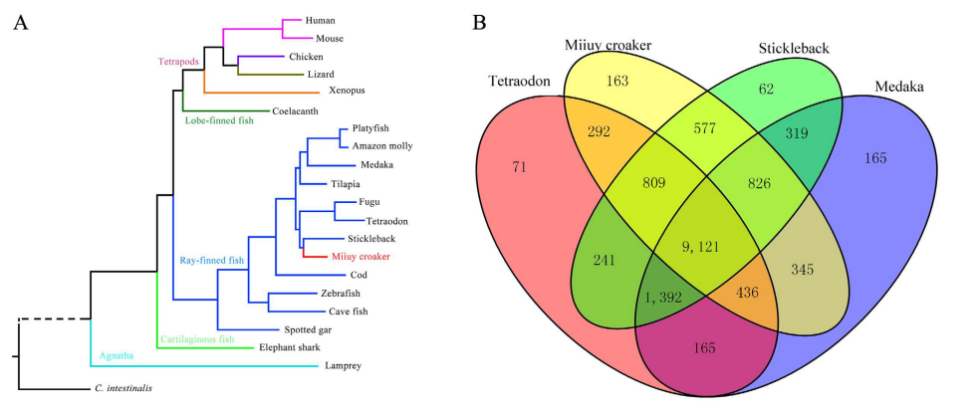
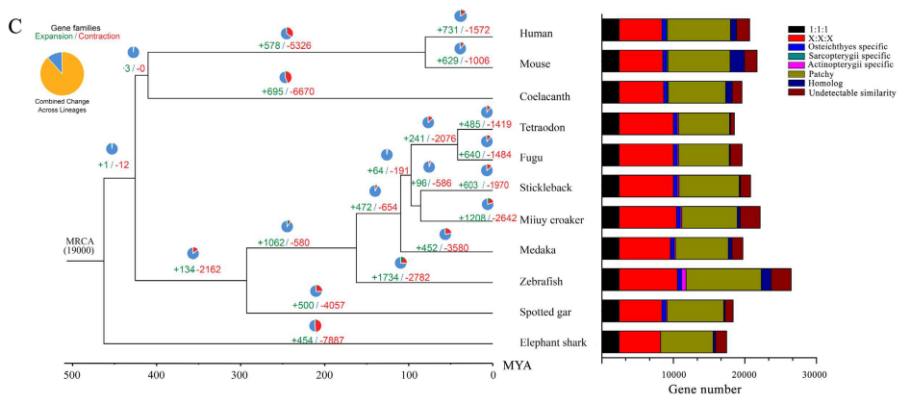
图1 系统发育关系分析 (A)21种脊索动物中,利用560个保守的单拷贝的同源基因构建系统发育树;(B)根据基因家族聚类分析,绘制四个物种(miiuy croaker, stickleback, medaka and tetraodon)的韦恩图,其中四个物种重合的部分表示必需基因(9,121),其余部分为非必需基因;(C)11种脊索动物的同源基因的进化和分布,蓝色和红色的数字分别表示扩增和缩小的基因家族,MRCA表示最近的共同祖先。
1. 鮸仔免疫系统的特征
鱼类含有很多种白细胞介素(ILs),包括IL-2, IL-4, IL-7, IL-9, IL-15和IL-21,这些ILs在获得性免疫应答中起到至关重要的作用,但是,在鮸仔中却只发现了IL-15。对于鮸仔基因组中与免疫相关的基因的研究发现,有两个MHC I基因和MHC II基因比其他测序的硬骨鱼要少很多(图2AB)。MHC基因家族的缩小以及与ILs相关的获得性免疫的缺失表明获得性免疫应答在鮸仔中是不起作用的。只不过相比获得性免疫而言,鮸仔拥有相当发达的先天性免疫系统。
肿瘤坏死因子(TNFs)是正常生理、免疫应答以及肿瘤退化过程中关键的先天性的细胞因子,研究者发现鮸仔中肿瘤坏死因子(TNFs)发生扩增,同时还发现了TNF α , 两个NFSF6 和三个TNFSF10,而且这些TNFs的含量要比其他鱼种的含量多。图2C的结果表明,鮸仔的特定区域有特殊的先天免疫;另外,在鮸仔中发现了50个NLR-C基因,这就表明,相比其他硬骨鱼而言,鮸仔的NLR家族发生扩增(图2D)。NLRs是最近发现的胞浆识别受体基因家族,在先天免疫应答过程中,主要负责识别病原体。图2E结果表明,鮸仔基因组中含有先天性免疫基因,比如干扰素 (IFN)和干扰素调节因子(IRFs)。因此,可以推断鮸仔发达的先天性免疫系统足以弥补它的获得性免疫的不完全发育。
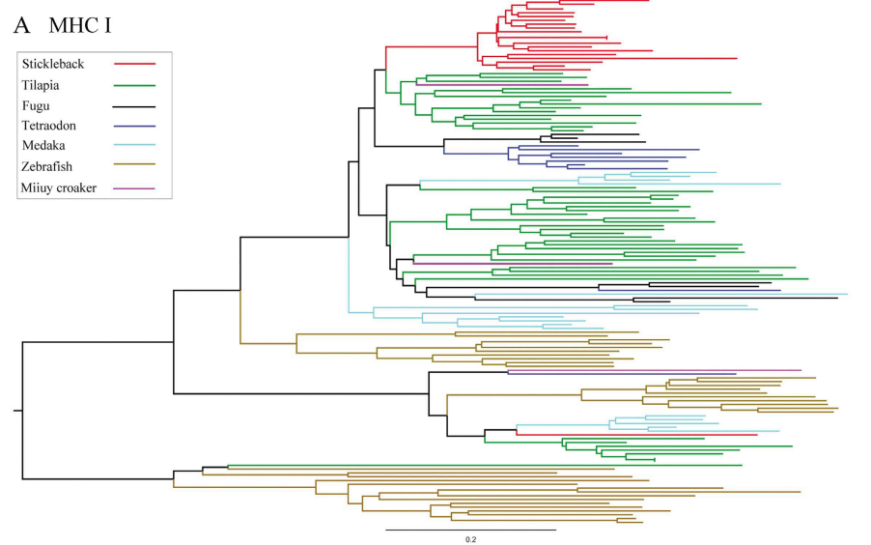
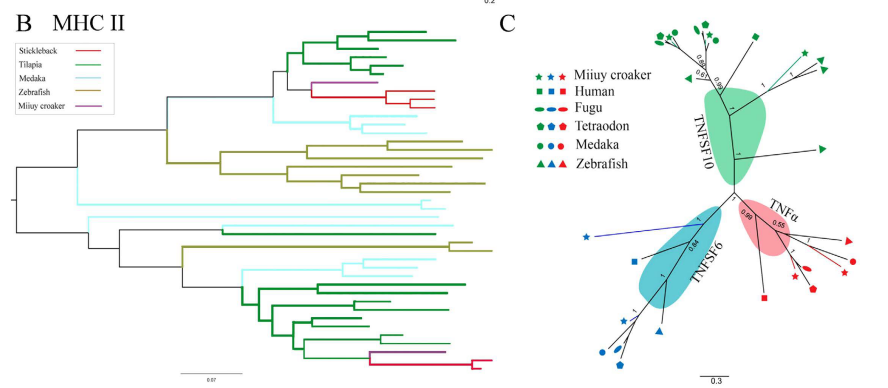

图2 鮸仔发达的先天免疫系统 (A)不同种硬骨鱼种,I类MHC蛋白复合物的系统发育树;(B)II类MHC蛋白复合物的系统发育树;(C)鮸仔和其他5种典型的脊椎动物中,TNF家族的系统发育树;(D)鮸仔和其他四类硬骨鱼的NLR-C家族的系统发育树;(E)鮸仔的免疫途径中,几个关键基因会发生改变,红色、灰色、棕色分别表示扩增的基因、缩小的基因和与其他硬骨鱼相似的基因。
1. 泥泞水环境下的感官适应
为了了解鮸仔在泥泞环境下的适应性,文章对视觉、味觉和嗅觉相关的基因进行了研究,并发现一些有利于鮸仔在泥泞环境中生存的特征。鱼类中发现有5种视觉色素基因,但是在鮸仔中只发现4种,即缺失了SWS1基因。SWS1是用来看紫外光(UV)的,鮸仔丧失了SWS1可能是由于浑浊的深海环境下并没有UV光。巧合的是,很少暴露在UV光下的深海鱼和夜行动物也同样缺少SWS1基因(图3A)。除了基因的缺失,SWS2基因的269位点的氨基酸(T, threonine)发生了重复和突变(图3B),A269T的替换转变了对于长波城区域的吸收波长,光谱宽度加宽增大了鮸仔视觉范围,这种转变也有助于鮸仔在浑水环境中衡量猎物以及掠食者的距离。
味觉是选择食物的重要因素,文章对鮸仔基因组的味觉感受器进行研究发现T1R2基因扩增了7次(图3C)。T1R2和T1R3会形成异二聚体T1R2/T1R3,其在哺乳动物中作为感受甜味的受体,但是在鱼类中是作为感受咸的受体。肉中主要是表达咸味的氨基酸,因此T1R2的扩增有助于鮸仔适应食肉的饮食习惯。同时还发现,肉食性鱼比杂食性鱼含有更多的T1R2基因。
为了进一步了解这种基因扩增现象,研究者又进一步做了系统进化分析。根据图3E的系统发育分析显示,鮸仔的T1R2s形成了一个与刺鱼关系较近的单系群。除了鮸仔和刺鱼,许多鱼为了适应水生环境也发生了T1R2基因的复制(图3C)。
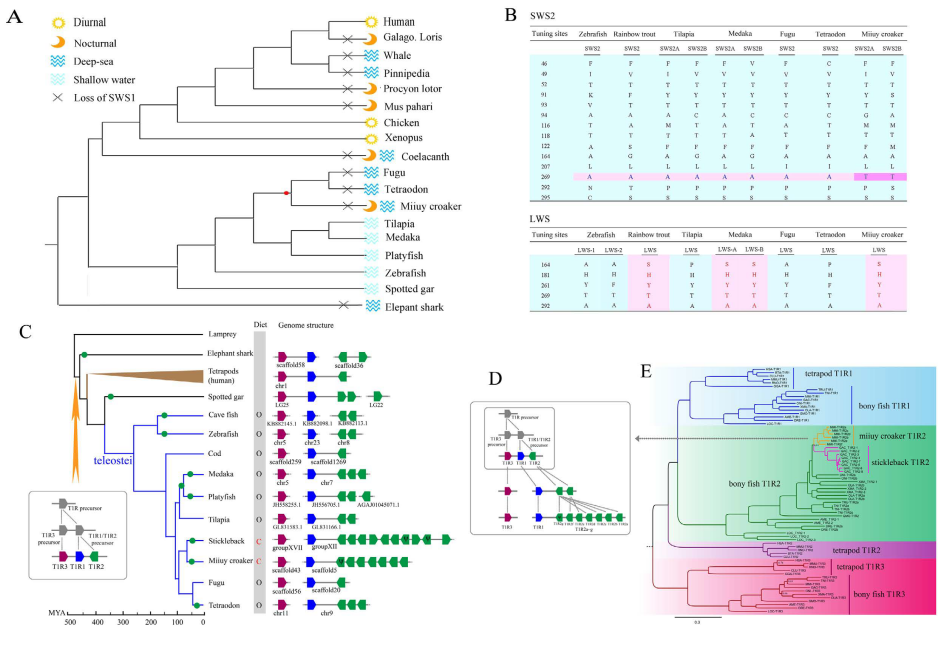
图3 鮸仔感官适应泥泞的栖息环境的遗传机制 (A)脊索动物中自然栖息环境与SWS1丧失的关系;(B)比较7种硬骨鱼中参与感光的蓝色红色视紫蛋白的氨基酸位点,并对这些位点的数字进行标准化处理;(C)硬骨鱼类(teleostei)中T1R2的扩增与栖息环境的关系,绿色的圈表示T1R2的复制,O表示杂食性鱼类,C表示肉食性鱼类,ψ表示伪基因;(D)鮸仔T1R2的进化假设,根据系统发生树推测T1R1和T1R2起源于共同的祖先,3个T1R基因起源于另外一个祖先;(E)鮸仔T1R2扩增的系统发育分析(隐藏了外群)。
【研究结论】
1. 文章成功完成了野生鮸仔基因组的高质量测序、组装和注释;
2. 对于鮸仔基因组序列的全面的分析促进了我们对于这类生物的基因组和进化水平的理解;
3. 研究者做了鮸仔的高质量的基因草图,发现几个对鱼先天免疫至关重要的基因家族;和其他鱼的基因组相比较,鮸仔的感官系统还是发生了一些变化,包括视力的改变以及味觉和嗅觉受体的改变,这些改变使得鮸仔在长期的自然选择中适应环境的改变;
4. 鮸仔的基因组研究可以为它的相对发育较好的的免疫系统以及在浑水中生存的适应模式提供可靠信息。
【所用到的软件和数据库】
Allpaths-LG:是一个基因组组装软件,适合于组装short reads数据,是现在行业内公认进行基因组De novo组装效果最好的软件。
SSPACE:这款软件可以利用Pair-End(PE)和Mate-Pair(MP)的二代数据将已有的contig或者scaffold连接成更大的scaffold,同时这个软件还能够利用PE或者MP的数据对已有的contig或者scaffold进行延伸。
Gapcloser:是用来对soapdenove或者其他软件在连接成scaffold过程中引入的gap进行弥补的软件,主要是借助read的成对关系。
Soapligner:用于快速高效地将短的寡核苷酸序列比对到参考基因组上。
SILVA:用来检查和比对RNA序列,既可以针对16S/18S,SSU,也可以针对23S/28S, LSU,包括了Bacteria, Archaea and Eukarya,同时也是ARB的官方指定数据库。
TRF:用来识别基因组中的串联重复序列的工具。
miRBase:是一个提供包括miRNA序列数据、注释、预测基因靶标等信息的全方位数据库,是存储miRNA信息最主要的公共数据库之一。
KEGG:是了解高级功能和生物系统(如细胞、 生物和生态系统),从分子水平信息,尤其是大型分子数据集生成的基因组测序和其他高通量实验技术的实用程序数据库资源,由日本京都大学生物信息学中心的Kanehisa实验室于1995年建立,是国际最常用的生物信息数据库之一,以“理解生物系统的高级功能和实用程序资源库”著称。