Radical remodeling of the Y chromosome in a recent radiation of malaria mosquitoes
疟蚊经过辐射后的Y染色体的重构
期刊:PROCEEDINGS OF THE NATIONAL ACADEMY OF SCIENCES OF THE UNITED STATES OF AMERICA (PNAS)
IF:9.423
年份:2016
【摘要】
在许多物种中,Y染色体都控制着雄性的基本功能特征,包括性别和生育。然而,由于Y染色体异染色质重复的抑制,使Y染色体的序列信息仅限制在少数的模式生物中,所以也限制了我们对于Y染色体在生命进化树的生物学意义的了解。文章对非洲最主要的疟蚊Anopheles gambiae进行长单分子测序,研究未发生重组的Y染色体的组成及结构。文章发现,An. Gambiae的Y染色体几乎完全由几个大量扩增、串联重复组成,还有一些可以与X染色体上相似的重复序列发生重组。经过辐射的种进行特异性别基因组测序,发现An. gambiae中序列发生迅速调整。对可以代表所有生长阶段的52个特异性别的An. gambiae的RNA-seq数据集进行分析,发现伴Y基因的一小部分缺少X同源的保留基因,但在其他除了An. gambiae的物种中是不伴Y的。所有种的Y染色体中,YG2是唯一的保守基因,但是在进化距离相对较远的蚊子基因组中,并没有发现与YG2相似的序列,表明这种传疟媒介中Y染色体基因进化速度很快。An. gambiae Y染色体的大量特征,为雄性蚊子的研究奠定了基础,对Y染色体的处理也有利于形成新的防蚊策略。
【研究思路】
取材:
样本来自An. gambiae的野生或者转基因G3种
文库构建:
基因组DNA库的构建使用了Pacific Biosciences recommended protocols和P4-C2 试剂
测序策略:
PacBio对基因组DNA进行三代测序
信息分析:
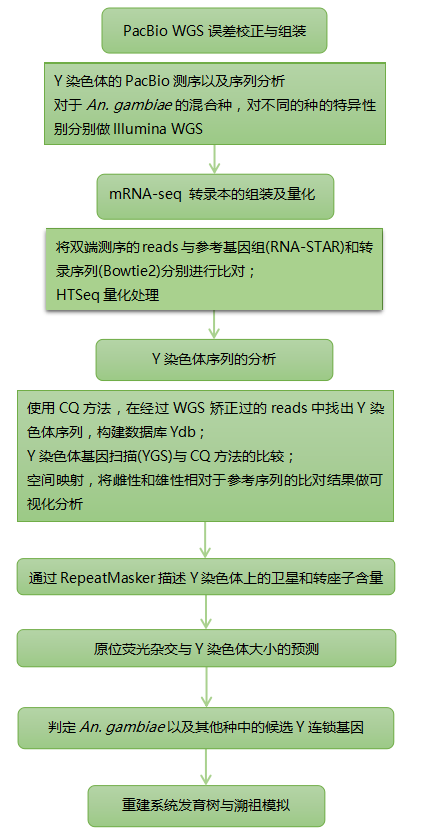
【研究结果】
1、 An. gambiae Y染色体序列的分析
为了对可能的Y连锁基因进行断定,研究者建立了两个计算方法,挖掘来自特异性别DNA池的short-read (Illumina)基因组序列。一种是Y染色体基因扫描法(YGS),另外一种是染色体商法(CQ)。CQ是根据序列与参考序列比对时的雌雄比例来推测是否Y染色体连锁,CQ法将未组装的reads分为一类,作为Y染色体序列数据库(Ydb)。图1显示,卫星AgY477是An. gambiae Y染色体的主要组成部分,并且有很强的雄性偏好性,在An. merus中也大量存在,但是并没有表现出强烈的性别偏好,而在An. arabiensis和An. quadriannulatus中并没有发现。结合这两种方法还发现8个基因(YG1-8),其中有三个是以前已经发现的。在An. gambiae所有的Y连锁基因中,只有YG2是在其他三个相近种中都存在的(图1)。
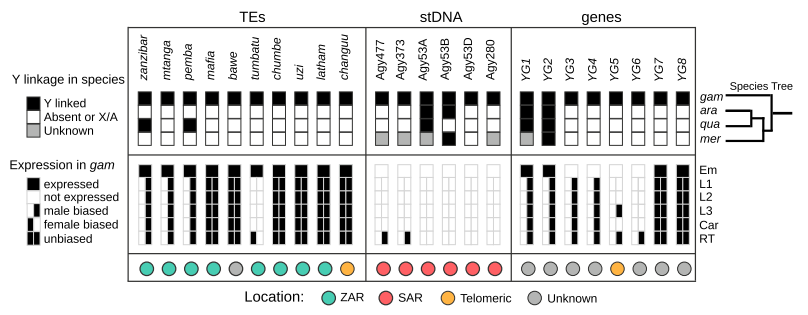
图1 不同种An. gambiae的Y染色体组成及表达模式 (最上面一行)黑箱子表示Y连锁,白箱子表示要么在不同种中完全缺失要么在它们的Y染色体中完全缺失,灰色箱子表示与Y连锁的未知状态。值得注意的是,灰色表示的序列的CQ或者RCQ的值大约为1,表明它们要么都在性染色体上,要么都在常染色体上。右侧是物种进化树,为Y染色体组成的变化提供了进化环境。在这4个种中,只有YG2基因是保守的。(中间一行)对特定性别的An. gambiae在不同生长阶段和不同组织(除胚胎)的转录本进行分析。(最下面一行)An. gambiae的Y染色体基因的组织结构。ara: An. arabiensis; Car:成年动物实体:Em:胚胎; gam: An. gambiae; L1–L3:第一到第三幼虫龄期; mer: An. merus; RT:成年生殖组织; stDNA:卫星 DNA; qua: An. quadriannulatus.
1、 An. gambiae的Y染色体包括大量扩增卫星和反转座子
图2A可以反映出Ydb中大约98%的碱基,数据显示NRY含有大量的同源的卫星DNA,Y染色体序列的主要特征,我们将之称作卫星扩增区域(SAR)(图2A)。图C显示不同的卫星DNA共同存在与相同的PacBio read中,这是由于姐妹染色单体或者染色单体内部发生交换而造成的。
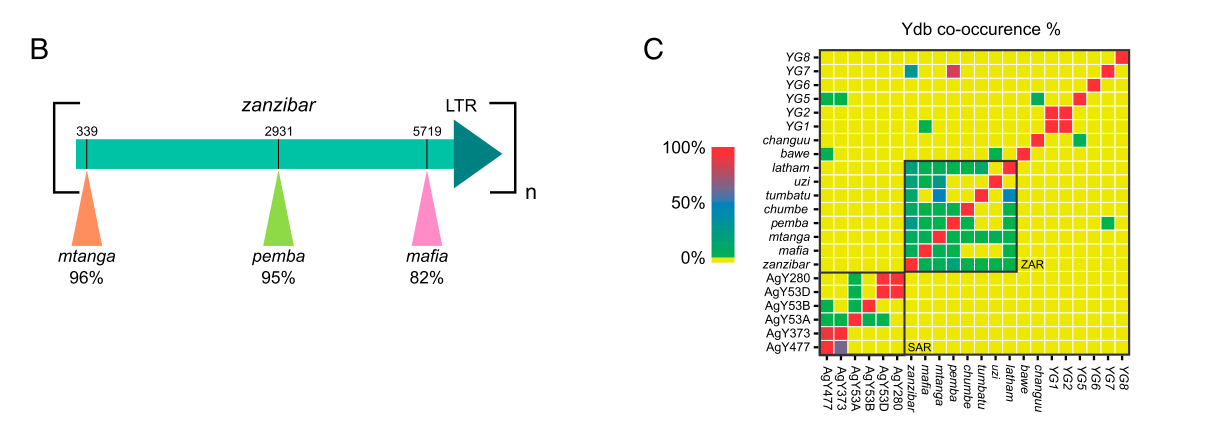
图2. An. gambiae的NRY(非重组的Y染色体)主要由一些卫星序列和转座子(TEs)的大量扩增串联序列构成。(A)An. gambiae Y染色体的两个主要部分(ZAR和SAR),在Ydb序列中占到92.3%,Ydb反映出An. gambiae中NRY的含量。左图是ZAR的组织结构,包含了zanzibar的从头到尾的串联排列,偶尔会有其他转座子的插入。每个zanzibar单元的黑绿色的箭头(B为放大图)代表单个的LTR,打断zanzibar单元的线表示其他TEs的插入。BAC10L19中的星号表示非典型的zanzibar单元(可能是由于重组或者错误装配造成的)。右图是SAR的组织结构,显示出每个同源单体序列(Y73915, Y46532, Y55593)。(B)是单个zanzibar单元的示意图,由多个domain和单个LTR构成。(C)来自Ydb的PacBio reads中Y染色体基因的共存在的矩阵。这些结果表明,卫星序列共存在于SAR中,TEs共存在于ZAR中,但是ZAR和SAR缺失相互独立存在的。
3、对幼年种辐射后Y染色体的动态结构变化
经CQ推测出的Y连锁结构,在An. gambiae的样本中都很相似,但是伴Y序列相应的拷贝数却是不稳定的(图3)。雄性样本中的卫星序列AgY53D和AgY280显示出SAR或ZAR成分的拷贝数的变化(图3)。YG2基因是唯一的保守基因,但是,却不是单拷贝基因,可能是受到比对到YG2的reads数量的影响(图3)。
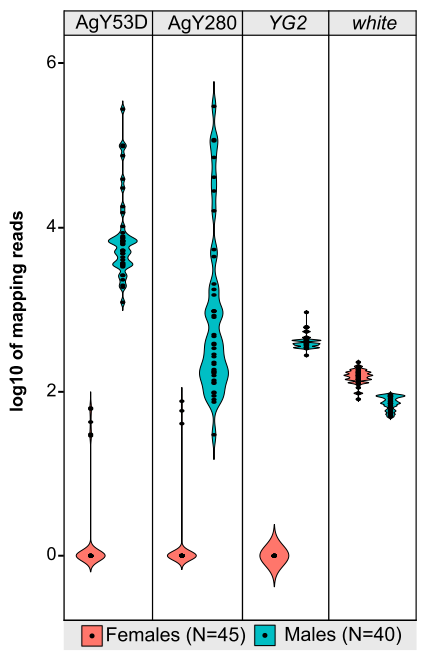
图3 卫星AgY53D和AgY280显示出An. gambiae自然群体中雄性的大量的结构活性,用小提琴图来表示,其中,蓝色表示雄性,红色表示雌性;YG2表示预测的决定雄性性别的基因;white表示单个复制的伴X基因。与YG2和white相比,个体间reads比对到卫星单体的数量差距很大。
为了研究种间的Y染色体结构变化的程度,研究者利用特异性别池的Illumina序列(Anopheles arabiensis, Anopheles quadriannulatus和Anopheles merus三个种)做FISH(荧光原位杂交)(图4),同时还对雄性偏好性以及相对丰度进行评估。
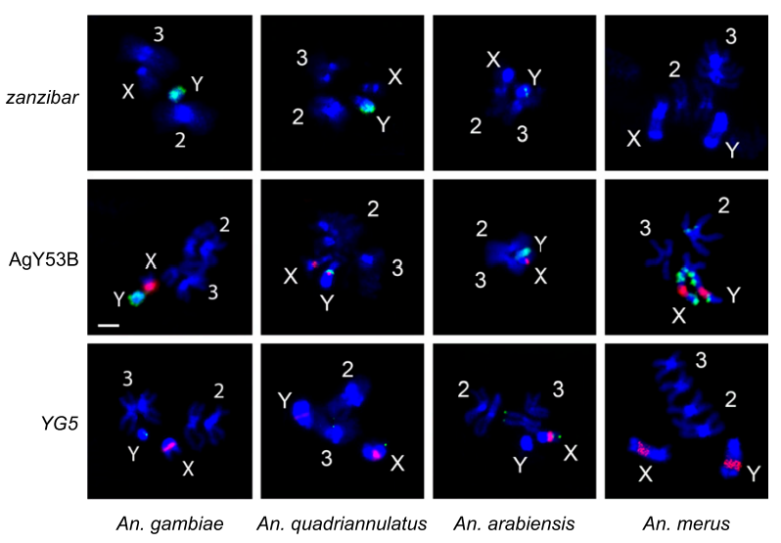
图4 物理图谱反应了An. gambiae及其他种的Y染色体序列的动态结构。在雄性An. gambiae Kisumu (zanzibar, YG5), An. gambiae Asembo (AgY53B), An.quadriannnulatus SANGWE, An. arabiensis Dongola, 和An. merus MAF种的染色体上做反转座子zanzibar, 卫星AgY53B, 和基因YG5 (绿色信号) FISH(荧光原位杂交)。除了zanzibar,所有试验都用到18S rDNA探针(红色信号),染色体用DAPI(蓝色)染色。
4、Anopheles属的XY染色体重组
对SAR的成分进行FISH(荧光原位杂交)映射(图4),或者通过显微剖析Y染色体荧光标记序列(图5A),都表明Y与X异染色质之间由于卫星单体的相似性而发生大量交叉重组。另外,文章还发现个别An. gambiae PacBio reads的重组印记(图5C),为之前的PCR扩增序列提供证据。
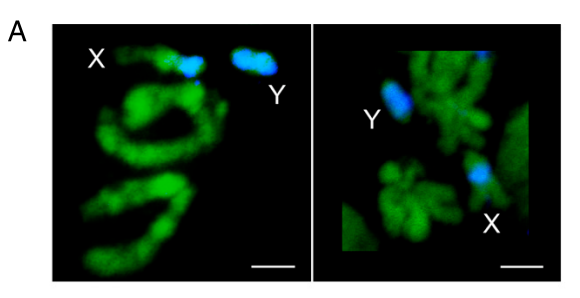
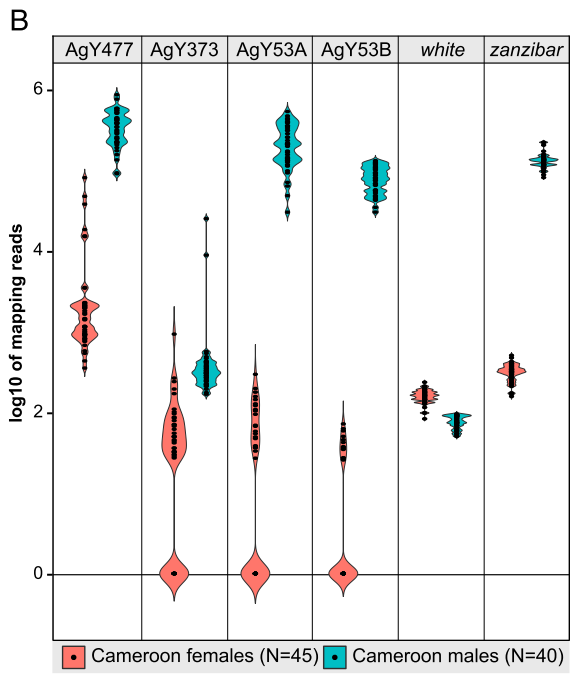

图5 An. gambiae的X和Y染色体并不是基因隔离的。(A) An. gambiae Pimperena种的雄性幼虫染色体分裂前中期(左图)和中期(右图)图,染色体使用YOYO-1(绿色)染色。(B)小提琴图显示的是来自An. gambiae 的40个雄性个体(蓝色)和45个雌性个体(粉色)的标准化后的reads比对到卫星AgY477, AgY373, AgY53A和AgY53B单体上的数量,并与比对到white gene (伴X单拷贝) 和 zanzibar(Y偏好)的数量进行比较。(C)两个PacBio reads的例子(pacbio_7224704_1和pacbio_5551309_1), 主要的伴X卫星(AgY367红色表示)和伴Y卫星(AgY373, 蓝色表示; AgY477, 黄色表示)都在图中表示出来。黑色箭头表示伴X和伴Y卫星的连接,紫色和橙色分别表示AgX367-AgY373以及AgX367-AgY477的重组,绿色盒子表示AgY477-AgY373的重组。
【研究结论】
1、 文章结合三代测序技术对对非洲最主要的疟蚊Anopheles gambiae进行长单分子测序,研究未发生重组的Y染色体的组成及结构。
2、 文章发现,An. Gambiae的Y染色体几乎完全由几个大量扩增、串联重复组成,还有一些可以与X染色体上相似的重复序列发生重组。
3、 对可以代表所有生长阶段的52个特异性别的An. gambiae的RNA-seq数据集进行分析,发现伴Y基因的一小部分缺少X同源的保留基因,但在其他除了An. gambiae的物种中是不伴Y的。
4、 An. gambiae Y染色体的大量特征,为雄性蚊子的研究奠定了基础,对Y染色体的处理也有利于形成新的防蚊策略。
【所用到的软件或数据库】
PacBio:是目前测序长度最长的测序方法,平均读长达到8kb。
RNA-STAR:对RNA序列进行快速全局比对,STAR, 很犀利, ENCODE专属RNA-seq工具. 在准度和时间消耗上, 效果拔群。
HTseq-count:是一款用于reads计数的轻便软件,可以用于多种mapping软件的输出结果。
ZenLite:一款成像软件,文中用来预测Y染色体大小。
Bowtie:Bowtie是一个超级快速的,较为节省内存的短序列拼接至模板基因组的工具。它在拼接35碱基长度的序列时,可以达到每小时2.5亿次的拼接速度。Bowtie并不是一个简单的拼接工具,它不同于Blast等。它适合的工作是将小序列比对至大基因组上去。它最长能读取1024个碱基的片段。模板最小尺寸不能小于1024碱基,而短序列最长而不能超过1024碱基。换言之,bowtie非常适合下一代测序技术;
Clustal Omega:在线比对工具。
PhyML:构建ML系统发育树的软件。