【文章题目】
番茄果实发育的单碱基分辨率甲基化图谱反映了成熟过程中的表观遗传修饰
Single-base resolution methylomes of tomato fruit development reveal epigenome modifications associated with ripening
【基本信息】
期刊:NATURE BIOTECHNOLOGY
IF:43.113
年份:2013
【摘要】
植物激素乙烯促进番茄果实成熟,但是一些未知信号却抑制了果实包括种子的成熟。为了判断这些信号是否包括表观遗传重塑,研究者用甲基转移酶抑制剂5-氮胞苷处理番茄,发现番茄提前成熟。文章对四个生长阶段的番茄果实进行全基因组甲基化测序,发现52,095个差异甲基化区域,此外, RIN(主要的催熟转录因子之一)结合位点定位在大部分催熟基因启动子的去甲基化区域,与去甲基化的发生基本一致。根据文章中的数据,发现表观基因组在生长的过程中并不是一尘不变的,建议在作物改良过程中,不仅要考虑到基因序列变异,还应该考虑表观基因组的编码信息。
【研究思路】
取材:
野生型番茄和Cnr与rin功能丧失的突变体均在温室标准条件下培育,取1个月的野生型番茄幼苗的叶片组织;在开花期之后,分别取17、39、42、52天的番茄果皮组织;取50 µL 1 mM1的5-氮胞苷溶液注射到未成熟果实的果轴中,对照组注射水,每周重复一次;当有早熟果实出现时,从经过5-氮胞苷处理的未成熟部分(绿色)和成熟部分(红色)分别提取DNA,用于测序分析
文库构建:
RNA末端测序建库:碱性磷酸酶对poly(A)RNA脱磷酸化,烟酸焦磷酸酶去掉5’帽子结构,连接酶连接Illumina 5’ small RNA接头,片段化处理后,加3’ small RNA接头,建库完成后经RT-PCR扩增并测序;
染色质免疫共沉淀测序:TruSeq index adapters用来构建Illumina ChIP-Seq库;
全基因组甲基化测序:利用全甲基化的Y-shape paired-end adaptor进行亚硫酸盐测序(BS-seq),用EpiTeck Kit做两轮的亚硫酸钠处理后,RT-PCR扩增
测序策略:
链特异性RNA测序:利用GAIIx或HiSeq2000完成;小RNA(sRNA)测序:sRNA加接头并经RT-PCR扩增后进行测序;全基因组甲基化测序:利用Illumina GAIIx Platform以及HiSeq2000 platform进行测序
信息分析:
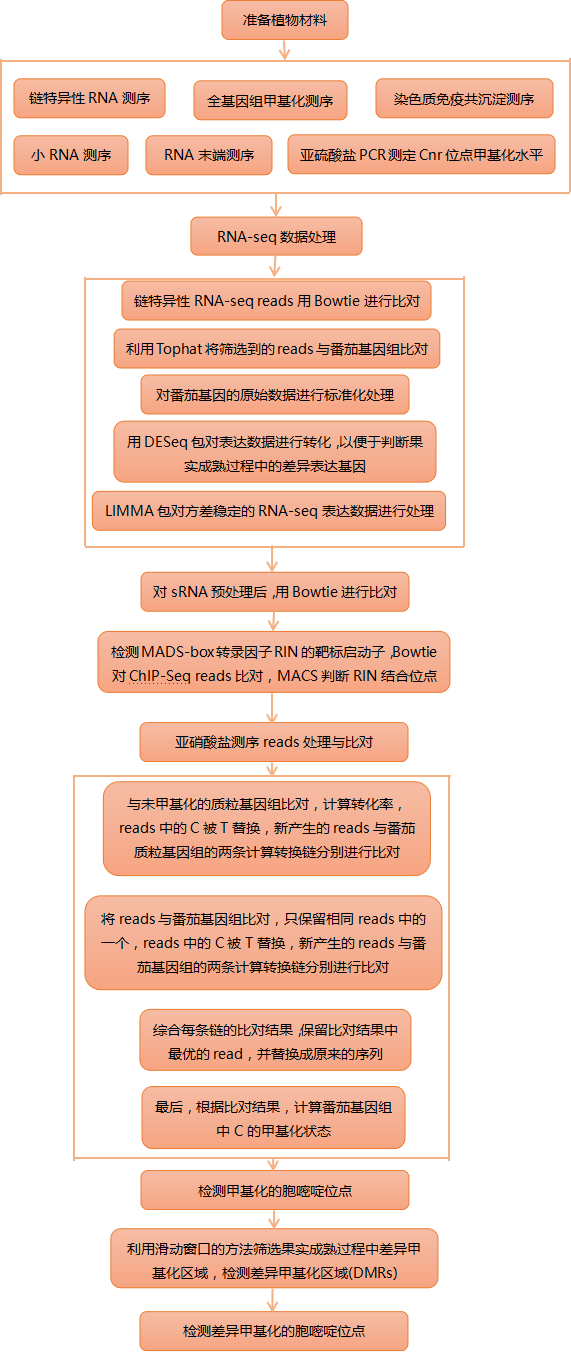
【研究结果】
1、甲基化转移酶抑制剂会促进果实提前成熟
研究者将5-氮胞苷注入未成熟的番茄中,观察与正常成熟的番茄的果实以及种子的差异(图1 a, b),接着,sanger亚硫酸盐测序证实,成熟果实红色部分Cnr基因5’上游区域发生去甲基化,但是绿色部分仍然保持高度甲基化(图1c)。同时,在果实早熟部分,文章还检测到成熟的标志基因的mRNA——pg2a和psy 1.
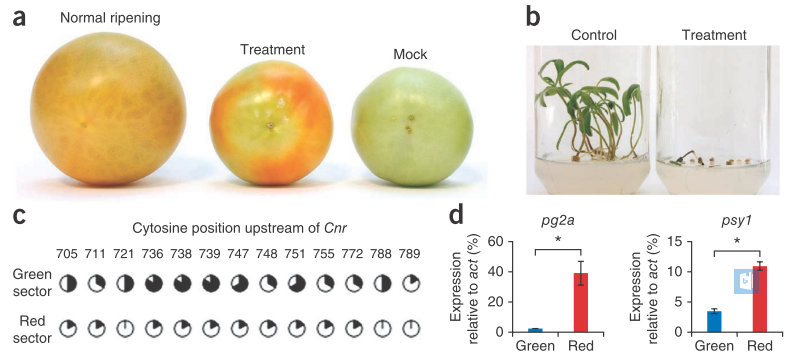
图一,甲基化抑制剂。在种子和果实成熟过程中的应用(a) 正常成熟左,5-氮胞苷处理(中)与水处理(右)得到的番茄果实;(b)经5-氮胞苷处理的番茄中提取种子做发芽试验(右),未做处理的番茄中提取种子做发芽试验(左);(c)经sanger亚硫酸盐测序,果实红色部分Cnr启动子区域去甲基化。数字表示相对于CnrORF起始位点的胞嘧啶的位置,黑色表示在指定位点胞嘧啶甲基化的频率;(d)相对于正常成熟Ailsa Craig turning果实,用5-氮胞苷处理的果实的红色与绿色部分所含成熟基因的差异表达。
2、番茄果实生长过程中甲基化测序及分析
番茄是世界上最大的被子植物,也是至今为止发现的唯一的果实中存在甲基化的作物。60%以上的基因包含高度甲基化的转座子,并且集中分布在异染色质的中心体附近(图2)。分布在染色体臂的常染色质区域,则含有较低的甲基化率以及甲基化密度(图2b)。
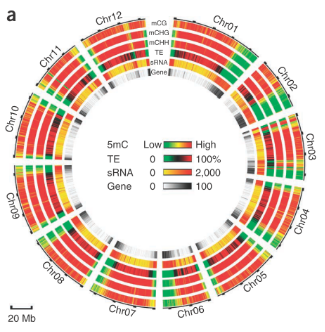
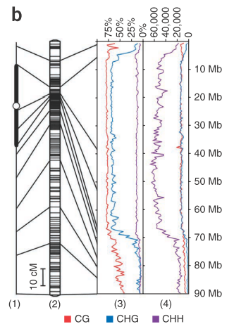
图2 番茄表观基因组研究 (a)序列中5-甲基胞嘧啶密度图,mC表示5-甲基胞嘧啶(H=A, C或T),TE表示转座子;(b)番茄Chr1的图谱,其中,水平衡线表示marker(标记)的位置,(1)表示染色质的位置;(2)来自Kazusa F2-2000连锁遗传图的番茄Chr1的基因图谱;(3)每个序列的DNA甲基化率;(4)每个序列的DNA甲基化密度。5mC, 5-甲基胞嘧啶; Chr, 染色体; cM, 厘摩; TE, 转座子。
3、番茄特定组织及生长过程中的表观遗传变异
研究者经亚硫酸盐测序后,发现特定组织和生长阶段,甲基化水平不同(图3 a, b)。例如,叶片中CG和CHG甲基化水平要比果实中高,但CHH相反。但是,从总体来看,这两个组织的甲基化水平还是很相似的。另外,全基因组甲基化水平的变化很大程度上反映了转座子甲基化反应的变化(图3c),这可能说明番茄基因组的表观遗传信息主要受到转座子丰度的影响。文章还发现,在野生型果实生长过程中,基因5’端甲基化水平逐渐降低(图3d),对野生型果实和叶片的甲基化信息做PCA分析发现,野生型番茄的四个生长阶段聚为一类(图3e)。
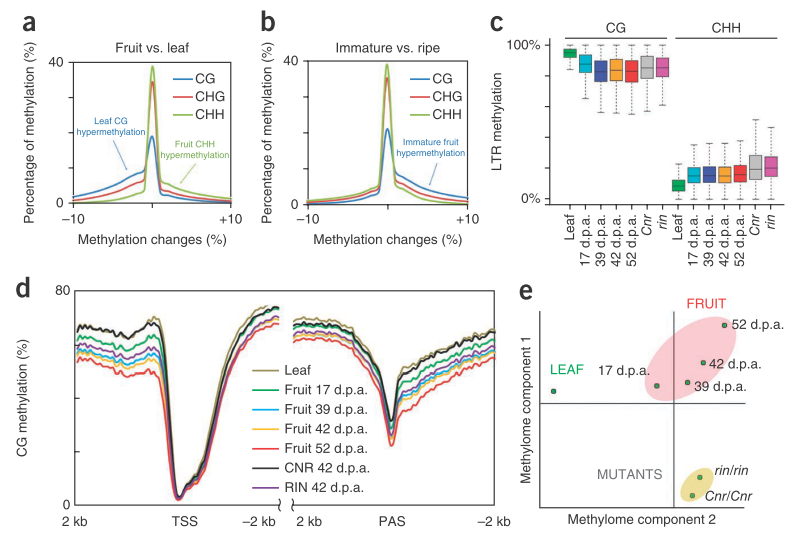
图3 番茄特定生长过程及组织的表观遗传变异 (a, b)番茄成熟果实与叶片(a)以及未成熟果实与成熟果实(b)之间胞嘧啶甲基化变异频率;x轴表示两个组织中平均甲基化水平的变动,y轴表示甲基化百分比;(c)在不同组织中,长末端重复序列(LTRs)的甲基化率;(d)基因中,GC甲基化率的变化,TSS:转录起始位点;PAS:多腺苷酸位点。(e)对野生型番茄叶片与果实以及突变型番茄果实的甲基化做主成分分析。
4、果实成熟过程中RIN结合位点的去甲基化
为了描述果实生长过程中表观遗传变异,文章利用滑动窗口的方法筛选野生型番茄四个不同生长阶段的差异甲基化区域(DMRs)(图4a),图4b反应的DMRs在染色体上的分布情况与最近的拟南芥的隔代遗传甲基化变异结果相似,但是,与拟南芥研究最大不不同之处在于,拟南芥甲基化变异主要分布在基因中间,而番茄的则主要分布在基因5’上游区域(图4c)。
此外,文章还研究了RIN结合位点附近的甲基化变化(图4d),发现所有组织中平均甲基化水平都低于RIN结合位点附近区域(图4e),RIN结合位点往往邻近DMRs(图4d)且在果实成熟过程中含有胞嘧啶的序列发生去甲基化,但是sRNA并没有发生变化(图4e)。
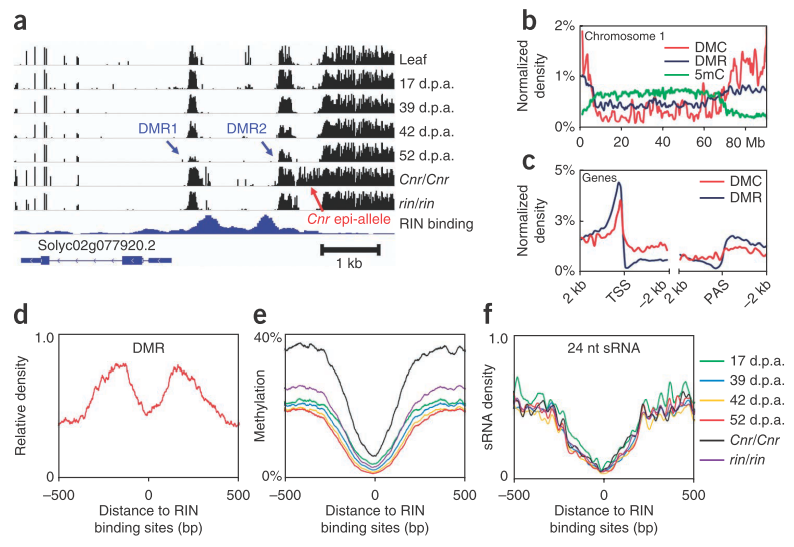
图4 果实成熟过程中RIN结合位点的去甲基化 (a)Cnr上DMRs和RIN的结合位点,蓝色箭头表示DMRs位置,红色箭头表示Chr表观等位基因的位置;(b, c) DMRs, DMCs和5-甲基胞嘧啶在Chr1(b)和基因(c)上的分布情况;(d-f)RIN结合位点附近的DMRs密度、甲基化率以及24nt sRNA密度。
5、果实成熟的调控模型
根据之前的研究,文章还提出果实成熟的调控模型(图5)。
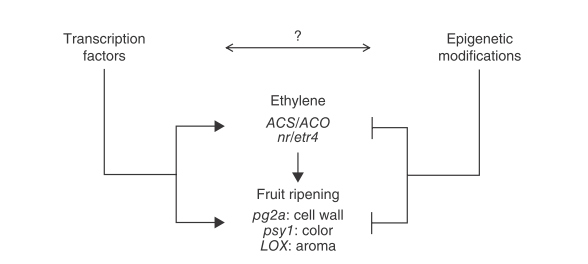
图5 果实成熟的调控模型 在不含能存活的种子的非果实组织以及未成熟果实中,是不会有果实成熟的,促进成熟的关键基因的启动子高度甲基化。在成熟的野生型果实中,它们的启动子发生去甲基化,并受到转录调控因子RIN和CNR的影响,但是机制尚不清除。转录因子的结合是否促进了启动子的甲基化,进而促进了果实成熟过程,尚不清楚。图中使用双向的箭头表示这个过程。但是,值得注意的是,早起使用抑制剂处理使启动子去甲基化,有效地促进了果实的早熟。
【研究结论】
1、 文章结合全基因组亚硫酸盐测序技术,展示出果实生长过程中大量的表观遗传特征,同时,对果实成熟过程中RIN目标启动子的特定位点的表观遗传变化做了相关分析。
2、 文章发现在rin丧失的突变体中,RIN结合位点发生高度甲基化,这与人类ENCODE计划和小鼠表观遗传研究结果相似。
3、 根据文章中的数据,发现表观基因组在生长的过程中并不是一尘不变的,建议在作物改良过程中,不仅要考虑到基因序列变异,还应该考虑表观基因组的编码信息。