Structural and Functional Analysis of the GRAS Gene Family in Grapevine Indicates a Role of GRAS Proteins in the Control of Development and Stress Responses
期刊:FRONTIERS IN PLANT SCIENCE
IF:4.495
年份:2016
【摘要】
GRAS转录因子参与植物生长发育的很多过程,还参与很多植物抗病以及非生物胁迫反应过程,但是在葡萄科中这类基因的研究却甚少。文章从最近上传的基因组中提取GRAS基因,构建基因模型并逐步改进,发现三个新的葡萄/木本植物特定基因。系统发育分析显示,GRAS基因被分为13个组,且被映射到19 V. vinifera染色体上。同时,还发现5个亚科,之前在其他种中并没有发现过。文章又通过多序列比对发现了典型的GRAS结构域并且还发现一个新的motif. 此外,对其他种的研究发现,无论是间断还是连续重复都会显著影响葡萄科GRAS基因的进化。不同组织在生物或非生物胁迫下的基因表达模式中,GRAS基因可能发挥不同的功能。通过对西红柿科和葡萄科的比较,在跃变期和非跃变期果实催熟过程中,发现一些保守转录因子的候选基因。总之,文章旨在提高多肉水果的质量,为进一步的功能分析提供相关候选基因等可靠信息。
【研究思路】
取材:基因序列来自之前报道过的GRAS蛋白编码基因、参考基因组来自URGI数据库、非冗余基因记忆COST注释基因来自ORCAE数据库、拟南芥GRAS蛋白序列信息从TAIR数据库获得、256个来自不同条件的基因表达数据来自三个不同的芯片平台(见测序策略)、其他16种植物的GRAS基因序列来自PlantTFDB数据库
文库构建:
测序策略:基因表达数据来自以下芯片平台:GrapeGen (21k probesets), Vitis Nimblegen array (29k probesets)以及内部的RNA-seq项目
信息分析:
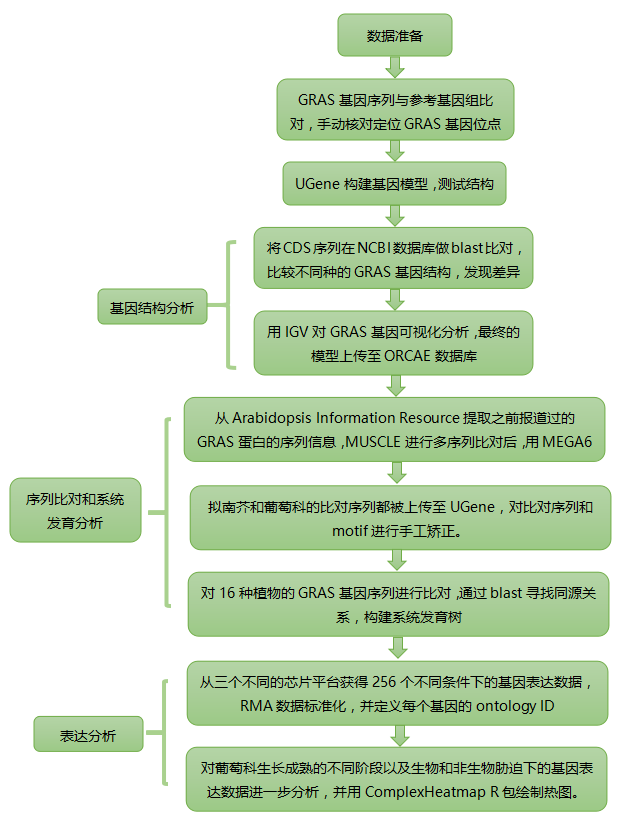
【研究结果】
1、系统发育分析
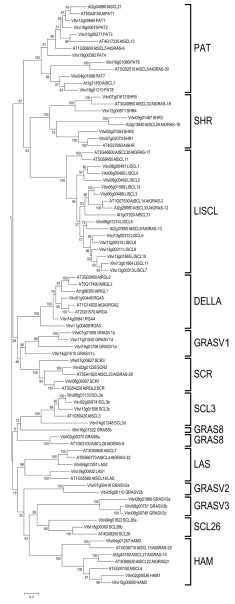
通过构建的系统发育树,发现了13个葡萄科的亚科,其中8个是已经发现的,另外5个是新发现的(图1)。有趣的是,有研究报道,西红柿科也有相同的13个亚科。文章还根据与拟南芥的同源关系以及基因特征对新发现的5个亚科进行定义。
图1 葡萄和拟南芥GRAS基因的系统发育分析。葡萄科中发现13个亚科:已经发现的DELLA, HAM, LISCL, PAT, LS, SCR, SHR, 和SCL3,还有新发现的5个亚科GRAS8, GRASV1, GRASV2, GRASV3, and SCL26.
2、GRAS蛋白结构及亚科特异motif
在GRAS蛋白C末端发现5个特征保守motif,分别是LHRI, VHIID, LHRII, PFYRE, SAW(图2)。图2还显示出不同的亚科所具有的特征motif以及保守motif:比如LHRI分为A和B两个单元,亮氨酸在A单元中反复重复出现;B单元包含一个核酸定位信号(NLS).
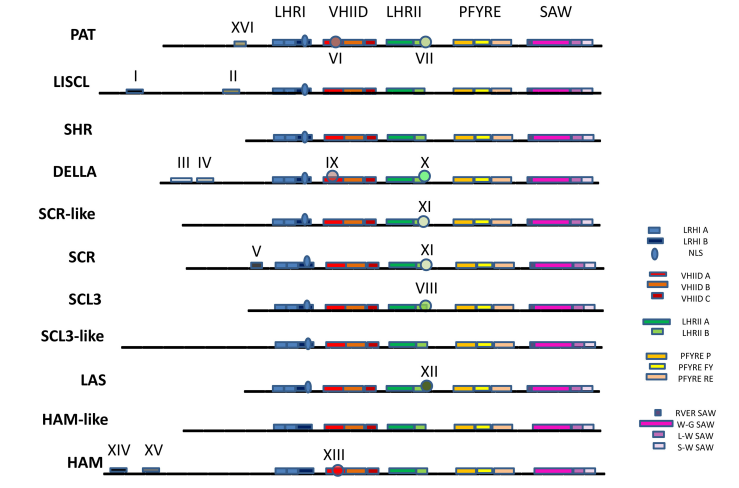
图2 GRAS蛋白结构和亚科特异motif. 上图显示5个保守motifs:LHR I, VHIID, LHR II, PFYRE, and SAW.亚科特异motif用罗马数字表示,GRASV1 (SCR-like) 没有结构域V. GRAS8 (SCL3-like)没有结构域VIII.GRASV2, V3, 和SCL26 (HAM-like) 没有结构域XIII, XIV, 以及XV. HAM和HAM-like没有NLS.
3、葡萄科GRAS基因与其他测序植物基因组的同源性分析
对V. vinifera葡萄科与其他物种的GRAS基因做同源性分析(图3),同源关系被分为三类:(i)出现在葡萄科但是不在其他物种中的基因;(ii)葡萄科基因与所给物种基因表现出一对一关系;(iii)葡萄科基因与给定物种存在同源性,但是没有明确的同源基因。图3反映出一些可能存在进化保守功能的基因,比如VviPAT1,VviSHR1,VviSCR1, and VviSCL26g,因为这些同源基因存在于给定的所有物种中。
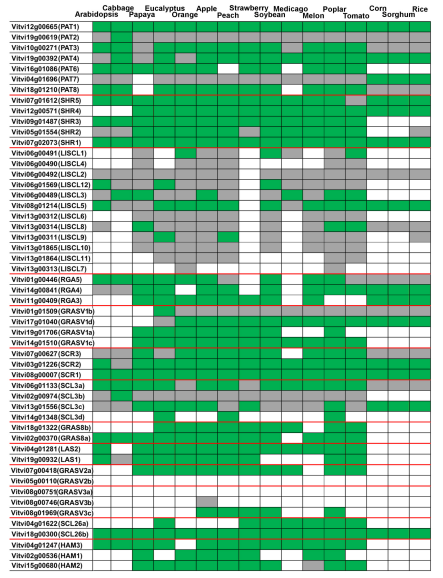
图3 葡萄科GRAS基因与其他测序植物基因组的同源性分析。绿色表示物种之间一对一的同源基因(最佳匹配);灰色表示葡萄蛋白和其他物种之间存在同源性,但是并没有检测到一对一的同源基因(不是最佳匹配);白色表示没有匹配。
4、GRAS基因在染色体上的定位
葡萄科基因组GRAS基因不均匀地分布在19条染色体上,crh 6和13染色体上GRAS基因数量最多,分别含有6和7个基因。同一组但分布在不同的染色体上的GRAS基因可能是由于祖先多倍体化产生了旁系同源片段。
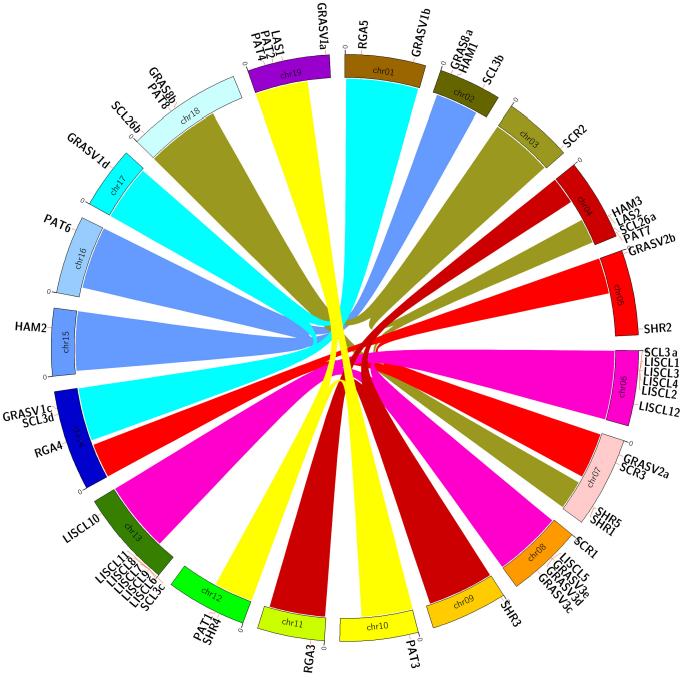
图4 葡萄科GRAS基因在染色体上的定位。其中,用相同颜色联系起来的不同染色体代表的是旁系同源区域。
5、葡萄科GRAS基因表达分析
基于公共数据库的表达数据的绝对值构建GRAS基因表达图谱,结果通过图5展现出来,发现VviPAT基因在所有的组织中都有表达,包括其他种。不同亚科的基因表达也不相同:对于SHR亚科,VviSHR1, VviSHR2和VviSHR3基因往往倾向于在除了花的器官和花粉粒以外的其他组织中;LISCL亚科的成员则表现出不同的表达模式,VviLISCL3, VviLISCL5, VviLISCL8和VviLISCL12在除了花粉粒以外的所有组织中表达,但是VviLISCL2, VviLISCL7, VviLISCL10几乎在所有组织中都不表达。VviRGA3, VviRGA4, 和VviRGA5基因在所有组织中都表达。VviSCR3在花粉粒、成熟果实以及衰老叶片中表现出表达高峰;还有三个SCL3基因表现出相似的表达模式。
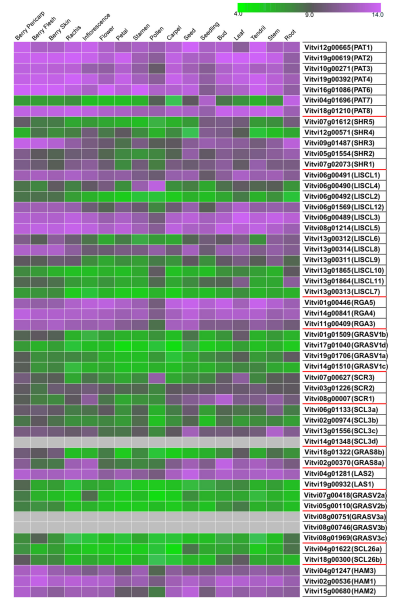
图5 葡萄不同组织中GRAS基因的表达。渐变的颜色表示经RMA标准化以后的表达强度值
6、葡萄生长成熟过程中、面对非生物胁迫,GRAS基因的表达
文章利用公共表达数据挖掘GRAS基因在果实成熟的过程中的特征。其中,VViPAT3, VviPAT4, VViPAT6序列相似度很高,由于受到成熟过程的影响,他们在开始成熟的时候表达量很低,而在过度成熟的时候表达量很高。文章发现另外一个有趣的现象,在Trincadeira和Corvina中,与AtSCL23直系同源的VviSCR2基因在成熟过程中表达下调(图6)。
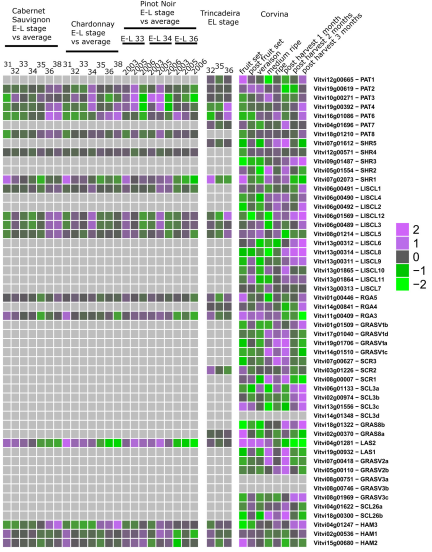
图6 葡萄生长成熟期GRAS基因的表达。左侧表示用GeneChip基因芯片实验;中间表示用Grapegen基因芯片实验;右侧表示用Nimblegen基因芯片实验。
除此之外,文章还对非生物胁迫和生物胁迫下,GRAS基因的表达作了分析(图7、8)。
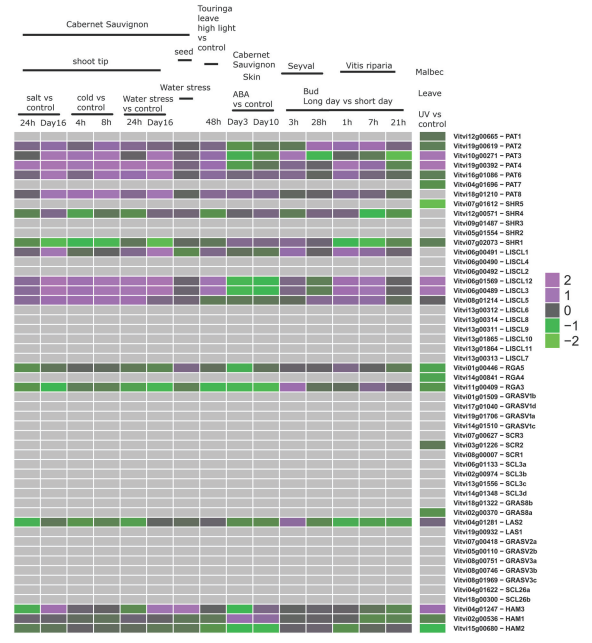
图7 非生物胁迫环境下,GRAS基因的表达。左侧表示用GeneChip基因芯片实验;中间表示用Grapegen基因芯片实验;右侧表示用Nimblegen基因芯片实验。
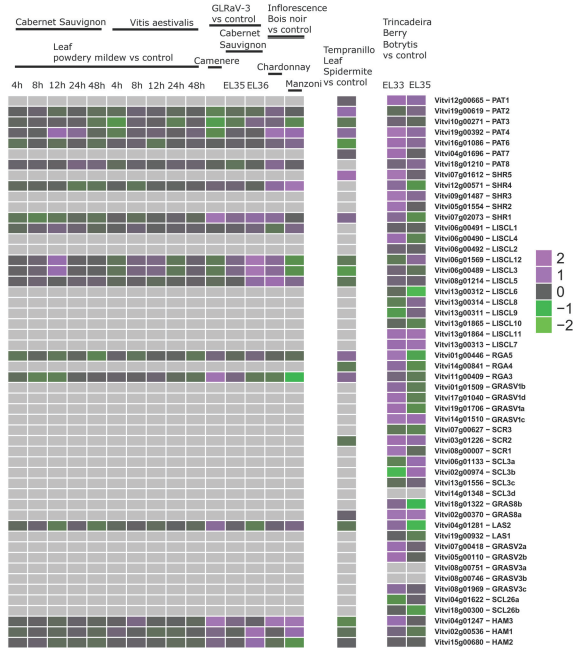
图8 生物胁迫环境下,GRAS基因的表达。左侧表示用GeneChip基因芯片实验;中间表示用Grapegen基因芯片实验;右侧表示用Nimblegen基因芯片实验。
【研究结论】
1、文章对葡萄科GRAS基因进行了详细的分析,包括(i)染色体定位;(ii)基因结构分析;(iii)与其他物种的系统发育及基因表达分析
2、文章通过系统发育分析,发现GRAS基因被分为13个组,且被映射到19 V. vinifera染色体上。同时,还发现5个亚科,之前在其他种中并没有发现过。
3、此外,对其他种的研究发现,无论是间断还是连续重复都会显著影响葡萄科GRAS基因的进化。不同组织在生物或非生物胁迫下的基因表达模式中,GRAS基因可能发挥不同的功能。
4、通过对西红柿科和葡萄科的比较,在跃变期和非跃变期果实催熟过程中,发现一些保守转录因子的候选基因。
【所用软件及数据库】
☆URGI数据库:预测植物或真菌的蛋白簇数据库( https://urgi.versailles.inra.fr/ ).
☆ORCAE数据库:提供在线的基因注释资源( http://bioinformatics.psb.ugent.be/orcae/ ).
☆UGene软件:用来构建基因模型,测试并修正基因结构。
☆IGV软件:是一种探索大型综合基因组数据的高性能交互式可视化工具。
☆TAIR数据库:拟南芥数据库( https://www.arabidopsis.org/browse/genefamily/GRAS.jsp ).
☆MUSCLE软件:用于多序列比对的软件。
☆MEGA6软件:用于构建进化树。
☆PlantTFDB数据库:植物转录因子数据库。